Tipp 46:
Über Codon Usage und Proteinexpression
Vergleichstool zur Codon Usage
Endlose Freuden der Molekularbiologie: Da findet der geneigte Forscher beim Blasten eine neue homologe Sequenz, bestellt sich die passenden PCR-Primer, produziert DNA, kloniert und ligiert – aber nach der letztendlichen Induktion findet sich nichts auf dem Western Blot, geschweige denn auf dem Coomassie-Gel. Woran liegt’s?
Seien Sie ehrlich: Die meisten von uns sind schon froh, wenn der Rechner am Morgen brav bootet und verweigern sich jeder tiefgehenderen EDV-Erkenntnis. Einige Spezialisten jedoch können erst nach einer intensiven Schrauberstunde glücklich einschlafen. Computer-Freak Thomas Schödl vom Institut für Pflanzenphysiologie und Zellbiologie der Universität Regensburg gehört zur zweiten Kathegorie. Vor kurzem hat er sich ein praktisches Online-Tool zur Visualisierung unterschiedlicher Codon Usages gebastelt:
"In-silico-Arbeit kann zwar nicht herausfinden, warum sich irgend ein elendes Protein nicht exprimieren lässt, aber sie kann – falls sich die Organismen phylogenetisch nicht allzu nahe stehen – zumindest einen Hinweis auf die Ursache des Problems liefern: die unterschiedliche Codon Usage nämlich. Wie aber kann man diesen Unterschied visualisieren? Zwar können etliche Bioinformatik-Tools Codon Usage-Tables erstellen, aber die sind meiner Meinung nach oft sehr nüchtern und unübersichtlich.
Um dieses Visualisierungsproblem zu lösen, habe ich drei Tools ins Netz gestellt. Mit deren Hilfe ist es möglich, Gene "Codon für Codon" oder als "Ganzes" mit einem anderen Codon Usage-Table zu vergleichen. Will mal nun ein humanes Gen beispielsweise in Hefe exprimieren, pastet man die DNA-Sequenz in das Textfeld und wählt als anzuwendenden Codon Usage-Table den von S. cerevisiae. Der Output sieht dann in etwa so aus wie auf der Abbildung ganz links.
In dieser Grafik lassen sich relativ schnell und leicht Sequenzen entdecken, die vom kumulativen Auftreten selten benutzter Codons geprägt sind. Möchte man den Überblick über alle 64 Codons auf einmal bekommen, so empfiehlt sich das zweite Tool: Hier wird für das Gen zuerst ein Codon Usage Table errechnet und dieser dann mit dem gewünschten Table verglichen (siehe Abbildung unten rechts).
Last but not least gibt es auch noch die Möglichkeit, einfach zwei Codon Usage-Tables zu vergleichen.
Aber das ist noch nicht alles: Es stehen zwar über 30 Codon Usage-Tables zum Vergleich zur Verfügung, aber andere, bei Kazusa (www.kazusa.or.jp/codon/) verfügbare können auch verwendet werden. Man surft auf der Kazusa-Website solange, bis man den "Wunsch"-Table gefunden hat und übergibt diesen Table an mein gcua-Tool, indem man die im Browserfenster angegebene Adresse kopiert. Als Output-Optionen steht neben html auch eine gut druckbare Ausgabe im pdf-Format zur Verfügung. Um die Grafen selbst zeichnen zu können, kann man sich die dazugehörigen Tabellen auch via E-Mail als Attachment schicken lassen. Wie oben schon erwähnt – experimentelle Probleme lassen sich so nicht lösen, denn auch in meinem Fall lösten sich die Expressionsprobleme erst nach Verwendung eines synthetisches Gens."
Zu finden sind die Tools übrigens unter folgender URL:
http://gcua.schoedl.de
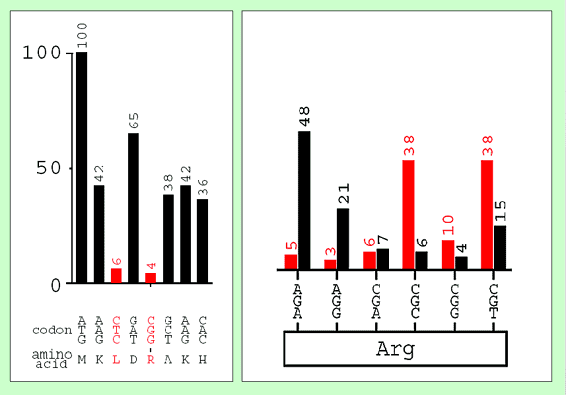
links: Das erste Codon ATG codiert für M(ethionin) und wird (nicht nur) von S. cerevisiae zu 100 % für die Codierung von M verwendet. Interessant wird es z. B. beim dritten Codon: Hier wird Leucin durch CTC codiert, aber S. cerevisiae verwendet dieses Codon nur zu 6 %. Ähnlich verhält es sich für die fünfte Position: Hier könnte ein Arg-Codon Probleme machen.
rechts: Die sechs Arginin-Codons werden in den Genen von E. coli und S.cerevisiae unterschiedlich häufig verwendet. E. coli verwendet für 76 % aller Arg-Codons entweder CGC oder CGT – in Hefe hingegen wird Arg hauptsächlich (69 %) durch AGA und AGG codiert.
|
Letzte Änderungen: 08.09.2004