Tipp 202: Thermoschalter für Proteinabbau
Bei vielen Experimenten wäre ein Schalter nicht schlecht, mit dem man Proteine gezielt an- und ausschalten kann.
Am Anfang der Arbeit an konditionellen Mutanten, die wir am Kölner Max-Planck-Institut für Pflanzenzüchtungsforschung begannen und am Leibniz-Institut für Pflanzenbiochemie in Halle weiter führten, stand der diffuse Auftrag die „Zellteilung temperatursensitiv“ zu machen. Hierzu wollten wir eine temperatur-empfindliche Zellzykluskinase in mutierte Zellen einschleusen, denen die Funktion der Kinase fehlte und die daher nicht wirklich lebensfähig waren. Unser Ziel war, die Zellteilung „ganz einfach“ über die Temperatur, wie mit einem Schalter an und auszuschalten: Warm = Zellteilung aus; Kalt = Zellteilung an. Eigentlich eine tolle Idee – aber wie sollten wir sie umsetzen?
Biologen verändern und steuern die Genexpression seit jeher, um die Funktion von Genen zu analysieren. In der Bäckerhefe, erzeugen sie die gewünschten Effekte zum Beispiel durch Mutationen, die die Zielproteine bei erhöhter Temperatur destabilisieren, während „kühlere“ Aufzuchtbedingungen stabilisierend wirken. Die Proteine funktionieren bei kälteren Temperaturen und werden im Idealfall nicht oder nur wenig durch die Mutationen beeinträchtigt. Man spricht deshalb auch von permissiven und restriktiven Temperaturen.
Schaltbare Allele
Um Stämme zu erhalten, die auf die Temperaturänderungen „antworten“ (responsive Stämme), muss man jedoch die entsprechenden Mutationen generieren und isolieren. Häufig handelt es sich hierbei um vordergründig unscheinbare oder sogar konservierte Punktmutationen. Dennoch können diese einen fundamentalen Einfluss auf die Funktion des Protein of Interest haben. Und sie lassen sich als schaltbare, konditionelle Allele – oft im Hintergrund einer sonst letalen Mutation – zur Analyse zellulärer Prozesse einsetzen, etwa im Rahmen einer konditionellen Komplementierung.
Sobald sich die Experimente von einzelligen Organismen wegbewegen, muss man sich anderer Methoden bedienen. Das gleiche gilt für Organismen, in denen eine Mutagenese viele Allele produziert (die man anschließend alle screenen muss) also zum Beispiel bei Insekten, Fischen oder Pflanzen.
Es gibt in diesen Fällen zahllose Methoden, die auf Gen-regulatorischer Ebene beispielsweise über induzierbare Promotoren funktionieren. Aber ganz abgesehen davon, dass das Schaltsignal im gesamten mehrzelligen Organismus wirken muss und Hitzestress oftmals physiologische Beeinträchtigungen auslöst, wenn er als Trigger verwendet wird, bleibt jedoch ein wesentliches Problem bestehen: Der Effekt spielt sich auf Ebene des Transkripts ab, und nicht etwa auf der des aktiven Enzyms oder funktionellen Proteins.
Gefährlicher Trugschluss
Das bedeutet im Klartext: Das Zielprotein dümpelt weiter in der Zelle herum und erfüllt seine Funktion, obwohl man es eigentlich „ausgeschaltet“ hat. Das Protein of Interest hat eine intrinsische Halbwertszeit und verweilt deshalb noch einige Zeit in der Zelle. Auch eine durchlässige („leaky“) Proteinbiosynthese, die auch ohne Aktivierung stattfindet, oder eine Restfunktion des eigentlich mutierten Targets können Ergebnisse verfälschen.
In Hefe löst seit 1994 die in Alexander Varshavskys Labor in Kalifornien etablierte temperaturgesteuerte Proteinabbau-Methode diese Probleme (Science 263:1273-6). Sie funktioniert über ein portables, also übertragbares temperatursensitives Abbausignal („Degron“), das mit den Zielproteinen fusioniert wird.
Zu selten genutzt
Leider wurde diese bahnbrechende Technik zwischenzeitlich nur einige Male in Hefe und in tierischen Zellkulturen verwendet und funktioniert auch nur oberhalb von 37° C oder 42° C (der restriktiven Temperatur des Hefesystems oder der Zellkulturen).
Das System, das aus dieser Zusammenarbeit letztlich entstand, basiert auf einer N-terminalen Fusionstechnik, die sich als Abbaupfad den N-end Rule-Pathway zunutze macht (Nat Commun 7: 12202). Die N-terminale Degron-Kassette besteht aus einem endständigen Ubiquitin und einer Dehydrofolatreduktase (DHFR), an deren Sequenzübergang (an Position 77) ein Phenylalanin oder Arginin positioniert ist. Beide Aminosäuren sind „destabilisierende“ Reste entsprechend der N-end Rule.
Übertragbares Abbausignal
Diese Aminosäure wird durch Enzyme des N-end Rule-Pathways erkannt und führt zur Poly-Ubiquitinierung des nachfolgenden Proteins. Um es noch komplizierter zu machen: Aus Aminosäure Nummer 77 wird Aminosäure Nummer 1, da wir eine sogenannte Ubiquitin-Fusionstechnik verwenden. Ubiquitin wird automatisch durch deubiquitinierende Enzyme cotranslational abgespalten. Die neue Aminosäure an Position 1 ist Phenylalanin oder Arginin und stellt im Zusammenspiel mit der DHFR das portable lt-N-Degron dar.
Wie kann man das lt-Degron in der eigenen Forschung einsetzen. Und macht es überhaupt Sinn, dies zu tun?
Zunächst sollte man überlegen, ob die durch eine leicht erhöhte Temperatur (25° bis 29° C) eintretende Destabilisierung (die je nach Protein Minuten oder Stunden dauern kann) mit dem Zielprotein kompatibel ist. Ist die „Schaltbarkeit“ notwendig, oder tun es auch die etablierten Methoden? Toleriert der verwendete Organismus den Temperaturshift? Kann ich unter 17° C kühlen, damit das Zielprotein akkumuliert und seine Degradierung über die N-end Rule behindert wird? Kann ich einen N-terminalen Tag von etwa 26 kDa an mein Zielprotein fusionieren, oder wird dadurch dessen Funktion, Lokalisierung oder Faltung negativ beeinflusst? Ist das Target in den für die N-end Rule und das Ubiquitin-Proteasomsystem (UPS) zugänglichen Zellkompartimenten, also im Kern und Zytosol zu finden?
Vorgefertigte lt-Degron-Kassette
Wenn nichts gegen die Verwendung des lt-Degrons spricht, können Sie das lt-Degron-Target-Fusionskonstrukt durch PCR oder Gateway-Klonierung herstellen. Die Details hierzu finden Sie sind im oben genannten Nature Communication Paper. Zudem haben wir die für die meisten lt-Degron-Fusionen benutzte K2-Kassette bei „Addgene” deponiert (pEN-L1-K2-L2; ID 80684).
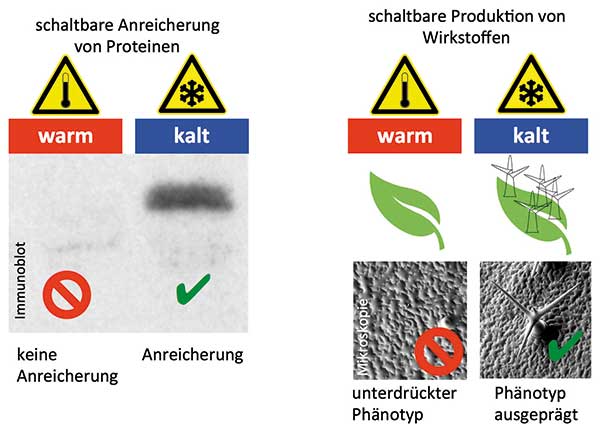
Das fertige Konstrukt baut man in einen Expressionsvektor ein und transformiert oder transfiziert damit die jeweiligen Zellen. Nach der Transformations- oder Transfektionskontrolle sucht man responsive Linien. Hierzu screent man die Zellen entweder auf geringe Level des Protein of Interest bei warmer Temperatur oder entsprechend hohe Level bei kalten Temperaturen. Die Ergebnisse verifiziert man jeweils bei der reziproken Temperatur.
Mittlerweile haben wir das lt-Degron erfolgreich in Arabidopsis, Tabak, lebenden Fliegen, Zellkulturen und Bäckerhefe (hier liegt die restriktive Temperatur bereits bei 17 °C anstelle der vormaligen 37 °C) etabliert. Zielproteine waren hierbei unter anderem Kinasen, Proteasen, Hydrolasen, sowie Transkriptionsfaktoren.
Nico Dissmeyer
(Nico Dissmeyer leitet die Junior Research Group am Leibniz-Institut für Pflanzenbiochemie in Halle).
Letzte Änderungen: 09.12.2016