Tipp 160:
siRNA-Pool mit Geißeltier-Häcksler
Alle Ausgangsprodukte in einen Topf schmeißen, dreimal umrühren und nach der Reaktion die hergestellten siRNAs isolieren. Simpler geht‘s nicht.
Eintopf-siRNAs
Kleine interferierende RNAs (siRNAs) sind ein beliebtes Werkzeug um Gene stumm zu schalten. Schleust man die etwa zwanzig Nukleotide langen siRNAs in die Zellen ein, werden sie vom RISC- Komplex zunächst zu einem siRNA-Einzelstrang entwunden. Dieser geleitet den Komplex zur komplementären DNA-Sequenz, die von RISC geschnitten wird.
Statt einzelne siRNAs gegen ausgesuchte Sequenzen zu synthetisieren, erzeugen viele Arbeitsgruppen einen ganzen siRNA-Strauß (siRNA-Pool), der das komplette Zielgen abdeckt. Dazu transkribiert man zunächst die beiden DNA Stränge des anvisierten Gens und erzeugt durch Annealing eine doppelsträngige RNA (dsRNA). Diese setzt man anschließend einer rekombinanten Variante des Enzyms Dicer vor, die die dsRNA zu siRNAs zerhackt.
Die Protokolle zur Erzeugung von siRNA Pools sind zwar einfach durchzuführen, beinhalten aber viele zeitraubende Einzelschritte. Keelan Guiley und Ashley Pratt vom Scripps Research Institut in Kalifornien, haben sich hierzu Gedanken gemacht und stellten kürzlich ein Protokoll vor, mit dem man siRNA Pools in einer simplen Ein-Topf-Reaktion herstellen kann (Nucleic Acids Research, 40, doi: 10.1093/nar/gkr1174).
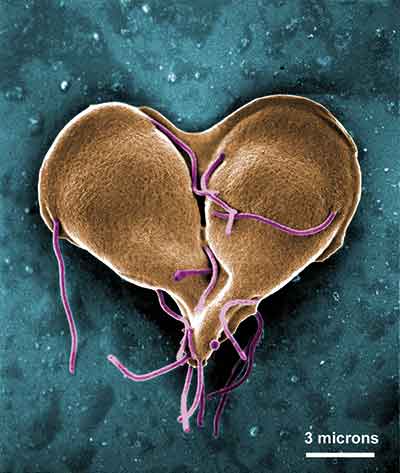
Schmalspur Dicer
Der entscheidende Trick ist, dass die beiden Kalifornier nicht das humane Dicer-Enzym benutzen, sondern eine Schmalspur-Variante aus dem Flagellaten Giardia lamblia. Dieser fehlen zwar etliche Domänen, die typisch sind für Dicer-Enzyme aus höheren Eukaryoten, dafür wartet sie mit einigen bemerkenswerten Vorteilen auf. So lässt sich Giardias Dicer-Enzym leicht exprimieren und reinigen und ist weniger anfällig für Produkthemmung als das humane Gegenstück.
Noch interessanter ist aber, dass Giardias Dicer-Enzym, im Gegensatz zu humanem Dicer, keine Probleme hat mit der Geschwindigkeit der in-vitro Transkription Schritt zu halten. Die neusynthetisierte RNA zerlegt sie dabei in 25 bis 27 Nukleotide lange siRNAs. Das ist zwar etwas länger als die 21 Nukleotide langen siRNAs, die humanes Dicer produziert, die längeren siRNAs werden jedoch von dem endogenen humanen Dicer-Molekül akzeptiert und auf RISC-taugliche Maße gestutzt.
Einfaches Protokoll
Entsprechend geradlinig und einfach ist die Ein-Topf-Reaktion zur Herstellung eines siRNA-Pools. Dazu amplifiziert man zunächst das Zielgen mit einer PCR. Die Amplikons dienen im nächsten Schritt als Transkriptionsvorlage für die zugegebene T7 RNA-Polymerase. Den Transkriptionsansatz ergänzt man mit einer adäquaten Menge des Dicer-Enzyms aus Giardia, das die synthetisierte RNA im gleichen Reaktionsansatz zu siRNAs zerhackt. Um die DNA-Template und sich eventuell ansammelnde RNA Einzelstränge los zu werden, setzt man nach dem Ende der Reaktion DNAse I und RNAse T1 zu. Im letzten Schritt reinigt man die hergestellten siRNAs schließlich und kann sie für die Transfektion von Säugerzellen einsetzen.
Giardias Dicer-Enzym kann man entweder selbst reinigen oder kaufen. Das kommerziell erhältliche Enzym ist jedoch, so die Ergebnisse von Guiley und Pratt, weit weniger aktiv als das frisch gereinigte. Die Inkubationszeiten verlängern sich dadurch von zwei bis drei Stunden für das frisch gereinigte Enzym auf etwa 16 Stunden. Es lohnt sich also, etwas Zeit zu investieren und das Dicer-Enzym aus Giardia lamblia selbst zu reinigen. Bewahrt man es im Gefrierschrank bei -80°C auf, bleibt die Aktivität etwa ein halbes Jahr erhalten.
Harald Zähringer
Letzte Änderungen: 31.05.2012