Protein-Schnappschüsse
Molekülbilder mit PyMOL
Kristian Rother
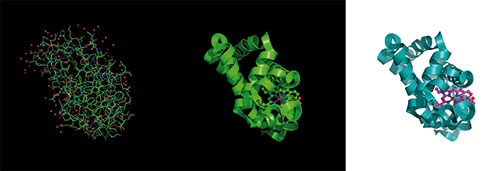
Aussagekräftige Bilder von Proteinstrukturen sagen meist mehr als fünf Seiten radebrechendes Wissenschaftsenglisch. Eine unentbehrliche Hilfe beim Knipsen von Proteinbildern ist das Programm PyMOL.
Wenn man ein Foto von seinen besten Freunden haben möchte, kann man jederzeit sein Handy zücken und auf den Auslöser drücken. Allerdings sollte man nicht erwarten, dass die Qualität der geschossenen Aufnahme für die Titelseite eines Hochglanzmagazins ausreicht. Hinter erstklassigen Fotos stecken meist viel Erfahrung und jede Menge Arbeit. Dies gilt nicht nur für Cover-Fotos von Zeitschriften, sondern auch für Bilder von Biomolekülen.
Die Bildkomposition
Wie aber kann man hervorragende Abbildungen in Publikationsqualität erstellen? Sicher nicht, indem man sein Molekül in ein beliebiges Programm lädt und anschließend auf „Bild speichern“ drückt. Zunächst muss man eine gute Bildkomposition, eine geeignete Darstellungsart und die richtige Perspektive finden.
Zum Glück gibt es dazu PyMOL, eine Bildmaschine für Moleküle, die viele der nötigen Schritte vereinfacht und bereits viele wissenschaftliche Artikel erfolgreich mit Bildern ausgestattet hat. Das Programm wird seit einigen Jahren kommerziell weiterentwickelt, die kostenlose Version 0.99 ist aber nach wie vor erhältlich (http://sourceforge.net/projects/pymol/ unter „all files“ und „Legacy“). Dieser Artikel stellt einen Fünf-Punkte-Plan vor, der die größten Hürden auf dem Weg zur gelungenen Molekülkalligraphie überwinden hilft.
Zuerst sollte man sich vergegenwärtigen, dass ein wissenschaftliches Bild eine klare Aussage haben muss. Was will man darstellen und warum? Anregungen dazu finden sich in einem Paper von Mura et al. (An Introduction to Biomolecular Graphics., PLoS Comput Biol., 2010; 6(8)). Erst wenn diese Frage geklärt ist, sollte man seine Strukturdatei laden.
Strukturen gibt es übrigens nicht nur in der Protein Data Bank (www.pdb.org), in der vor allem Proteine, DNA und RNA zu finden sind, sondern auch in der Pubchem (http://pubchem.ncbi.nlm.nih.gov/), die Kleinstrukturen von Metaboliten und Chemikalien bereithält.
Der für Anfänger schwierigste Teil ist das Auswählen gewünschter Teile wie Proteinketten, einzelne Reste oder Liganden. Aminosäuren, Nukleotide oder Kleinstrukturen bekommt man mit der Sequenzanzeige (das kleine [S] rechts unten) schnell in den Griff. Kompliziertere Operationen auf Atomebene sind mit dem „Select“-Befehl möglich. Dieser gleichzeitig mächtigste Befehl der eingebauten Skriptsprache erfordert ein wenig Übung, hat aber den Vorteil, dass man auf die ausgewählten Stücke jederzeit wieder zugreifen kann.
Protein-Cartoons
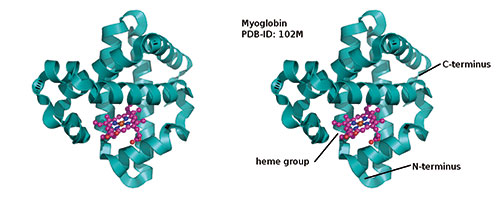
Für jeden Teil des Moleküls muss man eine Darstellungsart wählen. Diese findet man im Menü rechts oben unter den Knöpfen „[S]how“ beziehungsweise „[H]ide“. Sekundärstrukturen von Proteinen lassen sich als „Ribbons“ oder „Cartoons“ darstellen (hier kann man „Cartoon arrow“, „Cartoon loop“ oder „Cartoon dumbbell“ ausprobieren). Cartoons von Nukleotiden werden ansehnlicher mittels „Set cartoon_ring_mode, 3“. Die Oberflächendarstellung „Surface“ ist nützlich, falls man ein elektrostatisches Potential oder die generelle Gestalt eines Körpers veranschaulichen möchte.
Kleinstrukturen sehen am besten als Stäbe („Sticks“) oder „Kugeln und Stäbe“ aus. Letztere erreicht man, indem man sowohl „Sticks“ als auch „Spheres“ anzeigt, und in der Konsole „Set sphere_scale, 0.3“ eingibt. Mit „Set valence, 1“ werden Doppelbindungen dargestellt (sofern in der Strukturdatei annotiert). In vielen PDB-Strukturen gibt es außerdem noch Wasser und Ionen, die man nicht unbedingt im fertigen Bild sehen möchte. Mittels „[H]ide nonbonded“ schaltet man alle diese vereinzelten Atome ab.
Sobald die Darstellung steht, ist es Zeit sich mit den Farben zu beschäftigen. Bei der Farbwahl hängt viel von der Hintergrundfarbe ab. Schwarzer Hintergrund sieht auf Bildschirmen meist eindrucksvoller aus, aber beim Drucken geht dieser Effekt oft verloren. Das ist insbesondere in schwarz-weiß der Fall, abgesehen davon, dass schwarzer Hintergrund sehr viel Toner verbraucht. Weißen Hintergrund kann man mit „Bg_color white“ auswählen.
RGB oder CMYK
Eine weitere Überlegung ist die Wahl zwischen RGB-Farben (für Bildschirme) und CMYK-Farben (für Farbdruck). Der entsprechende Schalter findet sich im Menü „Display -> Color Space“. Die Umsetzung der CMYK-Farben ist in der Praxis oft schwierig, da sie letztlich vom Drucker abhängt. Als Faustregel gilt: Je dichter eine Farbe einer Primärfarbe ähnelt, desto sicherer ist es, sie zu verwenden. Viele der in PyMOL vordefinierten Farben lassen sich problemlos mit CMYK-Druck nutzen.
Spätestens in dieser Phase lohnt es sich, das Erreichte zwischenzuspeichern. Das Speichern als PyMOL-Session (.pse) sichert die geladenen Strukturen, die Kameraperspektive, die Farben und alle anderen Einstellungen.
Erst am Ende wird die Szenerie (besser gesagt: die Kamera) in eine aussagekräftige Perspektive gedreht. Dann wird der Auslöser gedrückt: PyMOL besitzt einen leistungsfähigen Raytracer, der eindrucksvolle Lichteffekte generiert. Mittels „Ray 3000, 2400“ wird ein hochauflösendes Bild erzeugt (in diesem Fall mit 3000 x 2400 Pixeln), welches mit „png abbildung.png“ gespeichert werden kann. Wie in der Fotografie gilt auch hier: nur wer viele Fotos macht, wird auch gute Fotos machen. Oft ist der schnellste Weg zum fertigen Bild, sein Molekül aus vielen Perspektiven zu „knipsen“, und hinterher das gelungenste Exemplar auszuwählen.
Feinschliff
Den letzten Schliff erhält das Bild durch sinnvolle Beschriftung und Symbole (Text, Pfeile, Kringel). Mit der PyMOL-eigenen Funktion kommt man hier nicht weit, die Beschriftung sollte man besser nachträglich mit Photoshop, PowerPoint, oder GIMP hinzufügen.
Der Rest ist Übungssache. PyMOL hat noch weitere Möglichkeiten zu bieten: Strukturen von Hand modellieren, geometrische Körper hinzufügen und sogar ganze Filme drehen. Ein ausführlicheres Tutorial zur PyMOL-Skriptsprache findet sich auf www.rubor.de/pymol. Damit steht dem eigenen Bild auf einer Titelseite nichts mehr im Weg.
Letzte Änderungen: 11.07.2012