Bilderflut
(13.11.17) Abbildungen aus wissenschaftlichen Publikationen kommen in den gängigen Suchmaschinen viel zu kurz. Die kostenlose Plattform SourceData soll das „Figure“-Meer bändigen, verknüpfen und zugänglich machen .
Ein Bild sagt mehr als tausend Worte – insofern es sich finden lässt. Denn das ist bei wissenschaftlichen Publikationen gar nicht so einfach. Die gängigen Suchmaschinen wie Pubmed und Google Scholar berücksichtigen häufig nur den Titel und das Abstract einer Veröffentlichung – nicht aber Diagramme, Tabellen und andere Abbildungen. Um diese zu finden, müssen die Paper meist mühselig durchforstet werden. Das raubt dem Wissenschaftler nicht nur kostbare Zeit, sondern auch Nerven. Außerdem besteht so die Gefahr, dass gar wichtige Ergebnisse übersehen werden, denn speziell in der Molekular- und Zellbiologie verstecken sich die wirklich wichtigen Informationen gerne in den aufwendig erstellten „Figures“.
Ein Meer aus Bildern
Bioinformatiker um den Molekularbiologen Thomas Lemberger von der European Molecular Biology Organization (EMBO) waren die mühselige Bildersuche leid. In Nature Methods stellten die Tüftler dieses Jahr ihre kostenlose Plattform SourceData vor (14: 1021-2). Sie soll es Forschern und Herausgebern ermöglichen, wissenschaftliches Bildmaterial wie Tabellen und Co so zu teilen, dass sie effektiv und intuitiv gefunden werden können. Dafür konvertiert SourceData Beschriftungen und Bildunterschriften in maschinenlesbare Metadaten, die anschließend in einer Datenbank gespeichert werden.
Einziges Problem: Gerade molekular- und zellbiologische Daten werden oft auf ganz unterschiedliche Art und Weise dargestellt. Abhilfe schaffen soll ein einheitliches Beschreibungsprinzip. Demnach werden zunächst alle biologischen Einheiten aufgelistet, die auf einer Abbildung eingetragen sind. Anschließend werden diese Einheiten einer von sieben Kategorien zugeordnet: Entweder handelt es sich dabei um ein kleines Molekül, ein Gen, ein Protein, eine Subzelluläre Struktur, einen bestimmten Zelltypen, ein Gewebe oder einen Organismus. Parameter wie Zeit, Gewicht oder Farbe sind erst einmal unwichtig.
Sobald die unterschiedlichen Einheiten eingeordnet sind, müssen ihre Beziehungen zueinander in einen Kontext gebracht werden. Aber von wem eigentlich? Aktuell erledigen das die Kuratoren der Journals, externe Kuratoren oder die Autoren selbst.
Zurück zu den Einheiten: Wichtig sind genau zwei Fragen: Welche Einheit wurde manipuliert (interventions), und welche daraufhin gemessen (assayed components)? Dieses System ermöglicht dem Nutzer sogar, nach einer spezifischen Hypothese zu suchen. Zum Beispiel: Wie beeinflusst Glucose das Hormon Insulin?
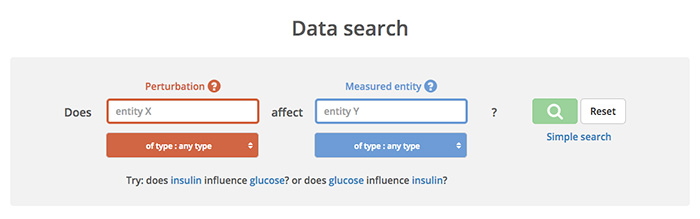
Eingabemaske bei SourceData: Was wurde manipuliert, was gemessen? (Quelle: https://search.sourcedata.io/)
Am Ende entsteht eine interaktive SmartFigure (siehe unten). Sie stellt nicht nur alle wichtigen Einheiten und ihre Rolle dar, sondern ermöglicht dem Nutzer, zwischen vorangegangenen oder anschließenden Experimenten hin und her zu navigieren. „SourceData verlinkt übergreifend verwandte Abbildungen zwischen Papern und Journalen und bildet dadurch einen durchsuchbaren Knowledge Graph, welcher durch Expertenkuratoren auf ihre Qualität geprüft wird“, erklärt Ko-Autor Robin Liechti vom Schweizer Institut für Bioinformatik in Lausanne in einer Pressemitteilung.
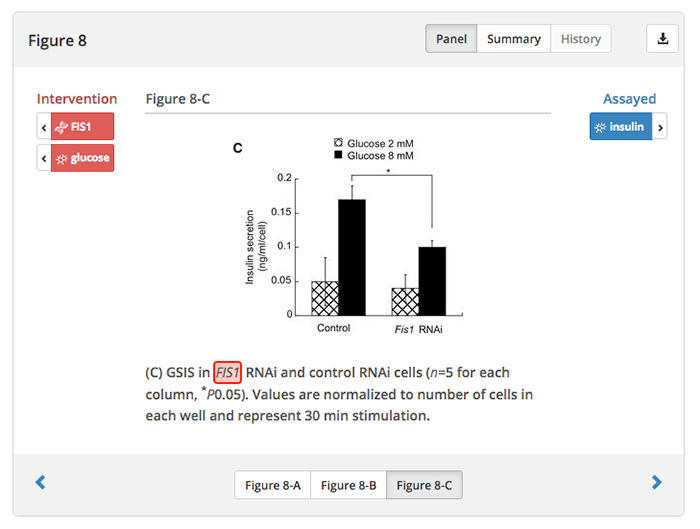
Auf einen Blick alle wichtigen Einheiten mit zusätzlichen Navigations-Möglichkeiten („Intervention“ und „Assayed“). Ein Klick auf „Summary“ zeigt,... (Quelle: https://search.sourcedata.io/panel/7502)
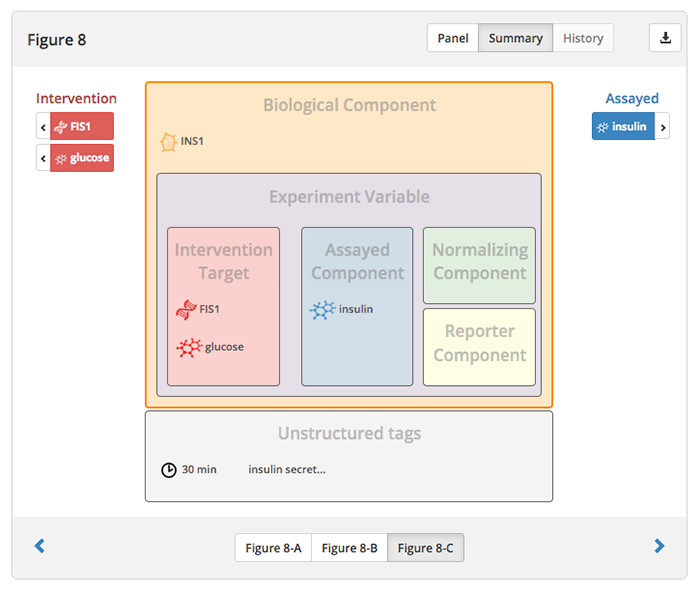
... welche Funktion die unterschiedlichen Einheiten in der Abbildung einnehmen. (Quelle: https://search.sourcedata.io/panel/7502)
Aber enthalten die Abbildungen inklusive Legenden alle Informationen, die für das Einspeisen in SourceData notwendig sind? Das testeten Liechti et al. an insgesamt 18.157 Abbildungen in 23 Journalen. Das Ergebnis: Bei 77 Prozent reichten die Angaben allein aus der Beschriftung in der Abbildung aus, um mindestens eine intervention/assayed components-Beziehung knüpfen zu können. In den übrigen Fällen mussten Kuratoren noch einmal in die Bildunterschrift schauen.
Ein Blick in den Text war nur nötig, wenn es um Fragen der Taxonomie ging. Diese ist dann wichtig, wenn beispielsweise ein bestimmtes Protein unter mehreren Synonymen bekannt sein sollte. Damit in solchen Fällen keine Verwirrung aufkommt, werden alle biologischen Einheiten mit einer standardisierten Taxonomie (zum Beispiel der NCBI Taxonomy oder Uniprot) verlinkt.
Aktuell beschränkt sich SourceData ausschließlich auf die Darstellung der Metadaten der Abbildungen – nicht aber auf die Interpretation der Ergebnisse. Das könnte sich jedoch bald ändern. Dann könnten Informationen aus dem Text oder Kommentare der Autoren zur Bildersuche ergänzt werden.
Juliet Merz
Alle Informationen noch einmal kurz zusammengefasst gibt es in folgendem Video: